基因表現是基因體上的 DNA 序列被聚合酶作用合成 RNA 的轉錄過程,整個轉錄過程中有許多不同的蛋白質共同參與,每個蛋白質複合體形成的環節都會影響轉錄的速度,假設降解速度相同的話,轉錄速度就直接影響轉錄量,也就是基因表現量。
染色體結構影響轉錄過程中蛋白質與 DNA 複合體的形成,直觀地來說,染色體還緊湊地纏繞的話,當然沒有聚合酶或其他相關蛋白質結合的可能。這種跟基因體序列內容無關的因素對基因表現的影響,稱作表觀遺傳調控。而跟基因體序列內容有關的因素又可以分成兩種調控類型,如果是緊鄰在基因上游的序列,因為序列特徵使其 DNA 雙股較容易解開或是可以被特定蛋白質結合,這種情況稱作 cis-acting,那段可以被辨識的序列叫做 cis-acting element,會辨識特定序列並且影響基因表現的蛋白質則叫做轉錄因子;相反地,如果影響的位置未知或是距離非常遠沒有特定距離關係的話,就會被稱作 trans-acting 的調控。
生物問題的困難之處就在於充滿變數,適度簡化問題才有可能逐步拆解想要了解的生命奧秘。從轉錄體分析中已經觀察到差異表現之基因,想要進一步溯源找尋可能的調控因素,除了查找共同表達的基因以外,可以嘗試由可能含有 cis-acting element 的啟動子 (promoter) 區段下手,大部分發表的研究會分析基因轉錄起始點上游 1500 bp 的序列當作啟動子區段。
cis-acting element 的分析流程非常簡單,將基因轉錄起始點上游 1500 bp 的序列掃描過去,很直觀地檢查是否有符合資料庫登記的 cis-acting element,且這個分析正反兩股都會偵測,畢竟在細胞核中沒有在分正反。線上文章常見提供植物啟動子相關研究的掃描服務有台灣成大出品的 PlantPAN2.0 、日本的 PLACE 和比利時的 PlantCARE,PlantPAN 對模式植物比較友善,PlantCARE 輸入序列之後需要等待 email 寄送結果,只有 PLACE 可以馬上拿到結果,因此我選擇 PLACE,拿取純文字的掃描結果在以自己的腳本進行視覺化。
在 PLACE 的主要頁面直接輸入序列,每次可以輸入 20 段序列
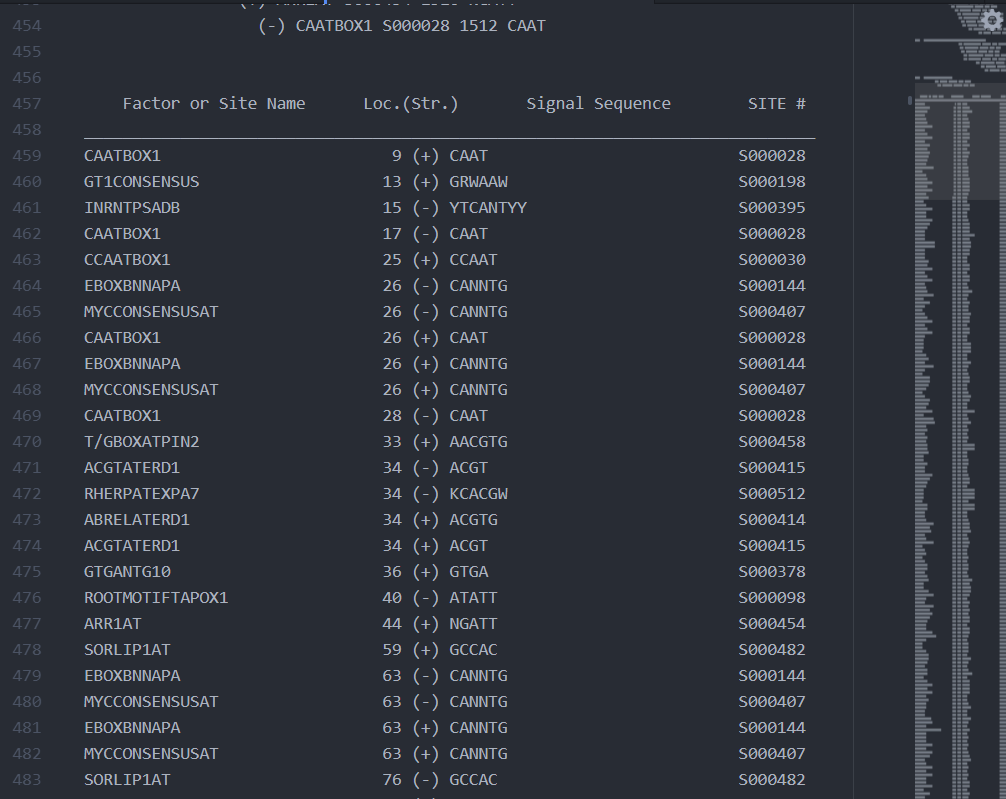
很快就可以得到一堆純文字的分析結果輸出,直接讀一讀就可以知道該段序列可能被甚麼轉錄因子結合或是受到甚麼生理環境影響,至於如何視覺化成更好理解一目了然的形式,將留待明天揭曉!
Plant cis-acting regulatory DNA elements (PLACE) database: 1999
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
